 您的购物车当前为空
您的购物车当前为空
为构建ERK5(MAPK7)激酶靶向化合物库,本研究采用已报道的4IC7和4B99PDB条目作为对接空间结构,使用薛定谔软件的Glide分子对接程序进行筛选。通过对两种对接模型(基于4IC7和4B99制备)的验证,发现4IC7结构能更准确地复现共结晶配体构象——配体ANP的晶体构象与最佳对接构象之间的均方根偏差值为0.6Å。随后从ChEMBL数据库中选取36个已知ERK5抑制剂(IC50<1µM)作为参考分子,用于对接程序的优化与验证。通过毒性筛选、不良基团过滤及PAINS过滤器对Life Chemicals库存化合物库进行筛选,基于通过过滤的化合物创建包含50,000个结构的多样性子集。
对接流程描述如下:
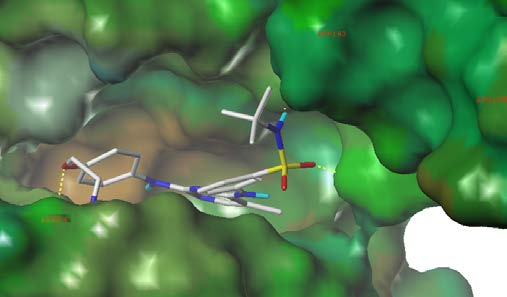
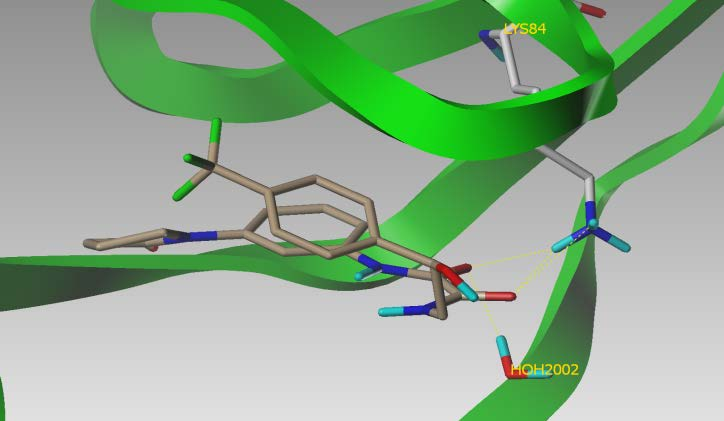
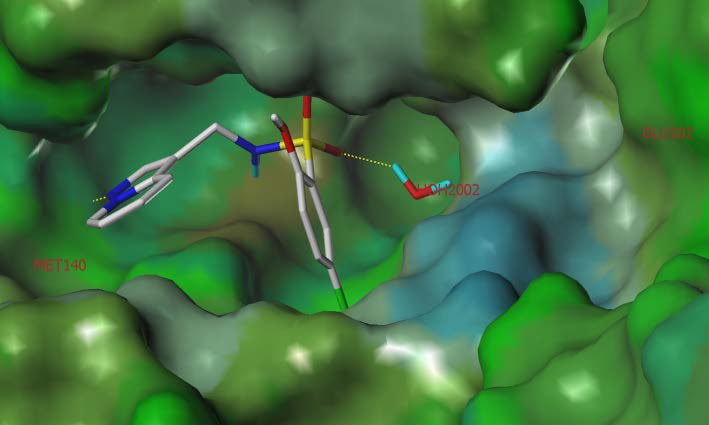
根据配体与结合位点特征确定了以下关键结合要素:配体中的嘌呤基团被识别为疏水(芳香族)特征;Met140、Asp200、Glu102、Gln146、Asp143、Arg98、Ala65、Lys84、Arg125、Asp138、Ser11等残基被确定为氢键作用位点;在默认参数下生成包含疏水性和活性残基信息的结合口袋静电图谱。随后采用从高通量(HTS)到标准精度(SP)的序贯对接筛选策略(图1)。
通过Gscore和Emodel评分函数对对接结果进行评估,筛选结果与已知ERK5抑制活性参考化合物的生物学数据高度吻合。最终预测约900个化合物对ERK5活性位点具有高结合亲和力(图2、3)。





 很棒
很棒

 |
|